Code-Performanz in R: Warum ist mein Code langsam
Dies ist der erste Teil unserer Serie über Code-Performanz in R.
Angenommen wir haben Code geschrieben, er läuft durch und er berechnet genau das, was wir brauchen - aber er ist unglaublich langsam. Wenn wir nicht dauerhaft bei unserer Arbeit ausgebremst werden wollen, müssen wir unbedingt die Laufzeit des Codes verbessern. Am besten findet man erst einmal heraus, wo man mit der Optimierung ansetzen sollte.
Oft ist nicht offensichtlich, welche Stelle den Code eigentlich so langsam macht oder welche von zwei Alternativen schneller ist. Dadurch entsteht die Gefahr, unnötig lange an der falschen Stelle herumzuoptimieren. Zum Glück gibt es viele Möglichkeiten, um systematisch zu testen, wie lang eine Berechnung läuft. Der schnellste und einfachste Weg ist die Funktion system.time. Man muss den Code nur in diese Funktion packen und schon erhält man - zusätzlich zu den Ergebnissen des Codes selbst - die Information, wie lang der Code gelaufen ist.
Ziehen wir zum Beispiel eine große Anzahl von Zufallszahlen:
system.time(runif(n = 1000000))
## user system elapsed
## 0.024 0.003 0.027
Der Wert unter "user" beschreibt (laut Dokumentation unter ?proc.time) "the time charged for the execution of user instructions of the calling process".
system.time ist allerdings nur geeignet, wenn die Berechnung etwas länger dauert (mindestens ein paar Sekunden). Falls eine Berechnung nur wenige Mikrosekunden dauert, ist das Ergebnis von system.time sehr ungenau und wird bei jeder Ausführung enorm schwanken. Es hängt dann zu stark davon ab, was das Betriebssystem zufällig in diesem Moment im Hintergrund tut. In diesem Fall müsste man den Code eigentlich viele Male laufen lassen und dann über die einzelnen Messungen den Mittelwert oder Median berechnen... Glücklicherweise tut das Paket microbenchmark genau das. Es führt einen Code-Block mehrere Male aus (standardmäßig 100 Mal) und berechnet dann einige statistische Kennwerte für die gemessenen Laufzeiten. Hier ein Beispiel, das zwei verschiedene Methoden zum Subsetting vergleicht:
library(microbenchmark)
microbenchmark(
"dollarSign" = iris$Sepal.Length[10],
"brackets" = iris[10, "Sepal.Length"]
)
## Unit: nanoseconds
## expr min lq mean median uq max neval
## dollarSign 584 651 1036.28 906.5 1042.5 15651 100
## brackets 8391 9054 11322.02 9355.0 9885.0 49936 100
Obwohl natürlich bei beiden Versionen dasselbe herauskommt, ist die Version mit eckigen Klammern im Mittel deutlich langsamer. Wenn wir jedoch einen Blick auf min und max werfen, sehen wir, dass wir im Extremfall zu einem anderen Schluss gekommen wären, wenn wir den Code nur einmal ausgeführt hätten!
Ein ganzes Skript zu optimieren, indem man stundenlang jede Funktion einmal mit system.time oder microbenchmark versieht, wäre wohl etwas umständlich. In diesem Fall kan man jedoch in RStudio auf den Profiler aus dem profvis-Paket zurückgreifen. Der Profiler analysiert die Laufzeit aller einzelnen Teile des Codes. Der Code muss lediglich in profvis() hineingepackt werden und dann einmal laufen. Dann öffnet sich die Nutzeroberfläche des Profilers und bietet einen schönen Überblick darüber, welcher Teil des Codes wie lang gelaufen ist.
Im folgenden Beispiel simulieren wir zunächst Daten, aus denen wir dann zwei Grafiken erstellen. Dann wird zweimal dasselbe statistische Modell berechnet, allerdings mit zwei verschiedenen Funktionen.
library(profvis)
profvis({
library(dplyr)
library(ggplot2)
# Daten simulieren
n <- 5000000
dat <- data.frame(norm = rnorm(n),
unif = runif(n),
poisson = rpois(n, lambda = 5))
# Berechnung weiterer Variablen
dat <- dat %>%
mutate(var1 = norm + unif,
var2 = poisson - unif + min(poisson - unif),
var3 = 3 * unif - 0.5 * norm)
# Grafiken
ggplot(dat, aes(x = var1, y = var3)) +
geom_point() +
geom_smooth(method = lm)
ggplot(dat, aes(var1)) +
geom_histogram() +
geom_vline(xintercept = 0, color = "red")
# Modelle
modLm <- lm(var1 ~ var2 + var3, data = dat)
summary(modLm)
modGlm <- glm(var1 ~ var2 + var3, data = dat,
family = gaussian(link = "identity"))
summary(modGlm)
})
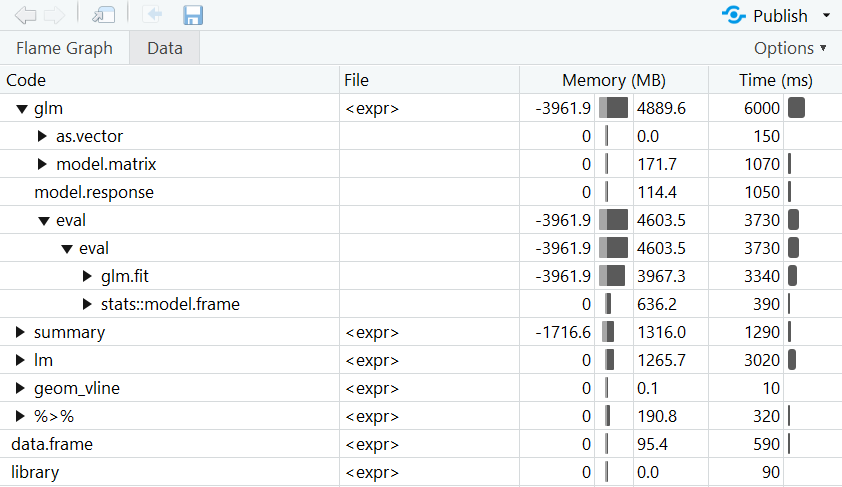
Hinweis: Die Berechnungszeiten schwanken stets aufgrund von Zufallseinflüssen. Deswegen variieren die Ergebnisse von system.time, microbenchmark und dem Profiler von Mal zu Mal.


